V okviru projekta TRANSyS objavljen členek v ugledni reviji Computational and Structural Biotechnology
Objava je nastala kot plod dolgoletnega uspešnega sodelovanja med Medicinsko fakulteto UL in Fakulteto za računalništvo in informatiko UL. Pri delu so sodelovali raziskovalci s Centra za genomiko in bio čipe, IBKMG UL MF, ter Laboratorija za računalniške strukture v sistemu UL FRI. Prvi avtor je Andrew Walakira iz Ugande, študent prvega letnika doktorskega študija Biomedicine in mladi raziskovalec na UL MF v okviru H2020 Marie Curie projekta TRANSyS.
Članek predstavi učinkovite protokole za izbiro genomskih presnovnih modelov, ki so ključni pri biološki interpretaciji podatkov transkriptoma. Predstavi tudi validacijo izbranega modela na realnih podatkih transkriptoma nealkoholne zamaščenosti jeter.
POVEZAVA DO ČLANKA: WALAKIRA, Andrew, ROZMAN, Damjana, REŽEN, Tadeja, MRAZ, Miha, MOŠKON, Miha. »Guided extraction of genome-scale metabolic models for the integration and analysis of omics data«. Computational and Structural Biotechnology Journal, ISSN 2001-0370, 2021, vol. 19, str. 3521–3530.
Povzetek:
Pojav visoko zmogljivih tehnologij je omogočil, da ustvarimo veliko količino različnih vrst omskih podatkov. To je omogočilo preučevanje organizmov na celični ravni. Vendar vemo, da življenje vzdržuje mreža reakcij, ki opredeljujejo biološki sistem. To pomeni, da preučevanje bioloških procesov na sistemski ravni daje boljše razumevanje osnovnih mehanizmov. Preučevanje bioloških sistemov in vivo je za nižje organizme pogosto zelo zapleteno, za višje organizme, kot so ljudje, pa celo nemogoče. To je posledica visokih stroškov in tudi etičnih razlogov. In silico so bili razviti računski modeli v obliki genomskih presnovnih modelov (GEM), ki zapolnjujejo to praznino. Podatki omskih raziskav so tukaj vključeni v referenčni GEM, da pridobimo kontekstno specifične modele, ki jih je nato mogoče uporabiti za proučevanje določenega stanja na sistemski ravni. Vendar pa je narava in zmožnost kontekstualnega modela v veliki meri odvisna od izbire algoritma za integracijo podatkov, znanega tudi kot metoda ekstrakcije modela (MEM). V tem delu uvajamo protokol za učinkovito pridobivanje kontekstno specifičnih GEM-ov na ponovljiv način. Poleg tega opisujemo pristope, s katerimi lahko analiziramo rezultate, pridobljene z izbranim MEM, in jih postavimo v biološki kontekst.
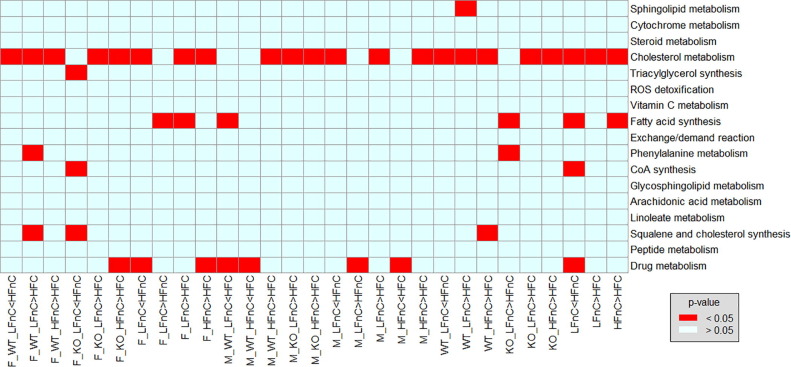
Slika predstavlja rezultat iz izbrane genomske metabolične mreže, in sicer obogatitve med posameznimi skupinami na različnih prehranah. Osredotočili smo se na podsisteme, ki so povezani s holesterolom. Model je pravilno napovedal, da je jetrna presnova holesterola obogatena pri miših na prehrani z visoko vsebnostjo maščob brez holesterola (HFnC), v primerjavi s prehrano z dodanim holesterolom (HFC). Primerjali smo jetra moških (M) in ženskih (F) miši divjega tipa (WT) in tistih, ki so razvile fenotip nealkoholne zamaščenosti jeter (KO). Nekatere živali so bile tudi na prehrani z nizko vsebnostjo maščob brez holesterola (LFnC).